刘元宁(1962),男,教授,博士.研究方向:生物信息学.E-mail:liuyn@jlu.edu.cn
siRNA与靶标mRNA的相互作用的热力学特征对于其干扰效率至关重要。RNAup是维也纳RNA软件包提供的预测靶标区域的可接触性(accessibility)的软件,将其预测的热力学特征加入到siRNA序列特征里,用支持向量机筛选高效的siRNA,该模型能够很好地预测出高效的siRNA,准确率达到88.12%,敏感度和特异性分别为88.35%和87.94%。
The thermodynamic of interactions between siRNAs and target mRNAs is crucial for the efficiency of its interference. RNAup is a software provided by Vienna RNA package, which can predict the accessibility of target region. By adding its predicted thermodynamic characteristics to the SiRNA sequence features and screening the efficient siRNA with support vector machines, the model can well predict efficient siRNA that the accuracy rate is up to 88.12%, the sensitivity and specificity are up to 88.35% and 87.94%, respectively.
RNA干扰(RNA interference,RNAi)是一种由双链RNA介导的、特定酶参与的基因沉默现象,1998年在秀丽隐杆线虫中进行反义RNA抑制实验时发现并命名,后来被发现是广泛存在的基因调节生物学现象[ 1]。siRNA介导的RNA干扰机制可以分为启动阶段和效应阶段。启动阶段是指RNaseIII核酶家族的Dicer与双链RNA结合,并将其剪切成21~23 nt及3'端突出的小分子RNA片段,即siRNA。效应阶段是指siRNA与若干个蛋白组成的RNA诱导沉默复合体(RNA-induced silencing complex,RISC)结合并解旋成单链,RISC被活化后,活化型RISC受已成单链的siRNA引导,序列特异性地结合在标靶mRNA上,并切断标靶mRNA,引发靶mRNA的特异性分解[ 2]。然而,并不是所有被设计出来的siRNAs都具有高效的干扰效率,针对不同靶标位置的干扰效率也不尽相同。基于此出现了一些所谓的高效siRNA设计规则,它们基本上都是基于siRNA序列特征的,比如:较低G/C含量,不宜形成复杂的自身结构(self-structure),在第三位最好是A。Ui-Tei等[ 3]提出了针对哺乳动物和鸡胚的高效siRNA四条设计规则:①反义链5'端为A/U;②正义链5'端为G/C;③在反义链5'末端三分之一富含AU;④不存在连续超过9位的GC区。虽然这些规则的机制并不完全清楚,但是这些规则已经被用来设计高效的siRNAs。在一项研究中,全基因组的siRNA被设计出来,通过人工神经网络用了大量的siRNA序列特征研究其抑制率[ 4, 5]。但是这些方法只考虑了siRNA的序列特征,不能全面覆盖影响其干扰效率的特征,比如蛋白的结合位点、细胞定位以及靶标mRNA的二级结构等。
已有大量的研究证明靶标mRNA的二级结构与siRNA的干扰效率有着重要的关系,特别是结合位点的局部二级结构对干扰效率有着至关重要的影响[ 6]。因此,RNA-RNA结合能的可靠预测是非常重要的。可以这样理解RNA-RNA的相互作用的热力学能量:①打开结合位点所需要的能量,这里有两个,分别是打开靶标结合位点的能量Δ Gm和打开siRNA的能量Δ Gs;②siRNA与mRNA结合所释放出来的能量Δ Gd。那么就可以得到siRNA与mRNA结合的总的自由能Δ Gt=Δ Gm+Δ Gs+Δ Gd。
本文中,利用RNAup预测siRNA与靶标mRNA相互作用产生的热力学特征,并结合一种全新的3-mer编码特征和传统的高效siRNA特征,通过支持向量机预测筛选高效的siRNA。
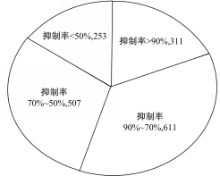
siRNA数据来自Huesken等[ 7]针对31条mRNA全基因组随机设计的2431条siRNAs,这些siRNA数据给出了它们的靶标基因的cDNA序列、siRNA精确的干扰抑制效率。由于目前RNA二级结构预测软件主要是针对长度不大于800个核苷酸的RNA序列(长度超过了800个核苷酸,预测准确率会急剧降低),因此为了提高mRNA二级结构预测的准确性,这里选择了针对23条长度小于800个核苷酸的靶标mRNA的1682组数据(见表1)。这些数据的抑制率分布见图1,其中抑制率大于90%的有311条,小于50%有253条。
| 表1 siRNA数据库及其靶标mRNA Table 1 siRNA library and targeted mRNA |
利用维也纳RNA软件包提供的RNAup WebServer来预测RNA-RNA结合热力学特征,通过输入siRNA序列和靶标mRNA(长度小于800 bp)即可得到打开靶标结合位点的能量Δ Gm和打开siRNA的能量Δ Gs以及siRNA与mRNA结合所释放出来的能量Δ Gd。则可得siRNA与mRNA结合的总自由能Δ Gt=Δ Gm+Δ Gs+Δ Gd。
本文中,共提取了与siRNA抑制效率密切相关的34个特征,主要分为以下3种:
(1)常规的siRNA序列特征,见表2,如siRNA的G/C含量、UI-Tei等[ 3]提出的4个哺乳动物siRNA设计法则等。
| 表2 常规siRNA序列特征提取方法 Table 2 General siRNA sequence feature extraction method |
(2)本文提出的3-mer编码序列特征,见表3,对siRNA进行重新编码提取出的3-mer序列特征20个,提取方法见图2,对siRNA进行两次重新编码提取出的20个3-mer序列特征,其中M代表碱基C/U,N代表碱基G/A;P代表碱基G/C,Q代表碱基A/U。
| 表3 3-mer编码序列特征提取方法 Table 3 3-mer coding sequence feather extraction methods |
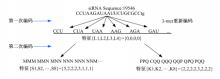 | 图2 对siRNA(编号为19546)进行重新编码提取出的20个3-mer序列特征Fig.2 Extracted 20 features of 3-mer motif from recoding siRNA(No.19546) |
(3)siRNA与靶标mRNA相互作用的热力学特征以及靶标的热力学特征,见表4。该特征为本研究首次应用到siRNA干扰效率的预测领域。这些包括RNAup预测的4个热力学特征Δ Gt、Δ Gm、Δ Gs以及Δ Gd,siRNA结合区域的位置P,其中P为第一个与siRNA结合的碱基的位置(从mRNA5'端记起)与mRMA长度的商。
| 表4 siRNA-mRNA结合热力学特征提取方法 Table 4 siRNA-mRNA binding thermodynamics feather extraction methods |
按照上述特征提取方法提取了所有1682组数据的全部特征信息,然后进行下一步支持向量机的数据学习训练。
LIBSVM[ 8, 9]的版本号为2.86-1,将1682组样本siRNA中抑制率大于89.5%的325组作为阳性样本,将小于54.9%的382组作为阴性样本,样本集大小为707。将样本的34个特征值输入到LIBSVB中,随机抽取500个作为训练集,剩下207作为测试集。采用交叉验证的方法在一定范围内寻找最佳参数 -c与 -g。重复训练10次取平均值。
本文采用五次交叉验证来寻找LIBSVM分类器的最优参数,每次实验都随机选取测试集和样本集,最后取10次实验平均值。分别计算出分类器的Accurary,Sensitivity,Specificity,MCC和Precision值:
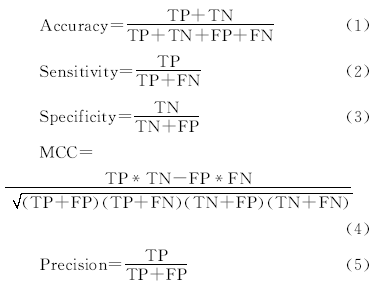
式中:TP代表分类器预测结果中真阳性的数目;TN打表真阴性的数目;FP代表假阳性的数目;FN代表假阴性的数目。
分类器的评价实际上就是对预测效果的评估。由式(1)~(5)可以看出,敏感度实际上就是真阳性率;特异性衡量的是该模型预测对个体特异性变化的应变能力。准确度就是预测正确的比例。这些参数能够很好地评测分类器的好坏[ 10]。
从表5可以看出,本文提取的20个3-mer编码特征和7个siRNA-mRNA结合热力学特征再加上7个常规的siRNA序列特征可以很好地进行高效siRNA分类筛选,分类器的预测准确率达到了88.12%,敏感度和特异性分别为88.35%和87.94%,MCC值为0.7609,Precision值为0.8593,通过对照实验(见表6)可以看出随着3-mer编码特征和siRNA-mRNA结合热力学特征的加入,分类器预测的准确率分别提高了6.19%和3.1%。并且分类器的其他评测参数也均有不同程度的提高,说明本文提出的siRNA-mRNA结合热力学特征和3-mer编码特征对siRNA干扰效率的预测是有效的。
| 表5 所有34个特征进行十次SVM训练得到的分类器的表现 Table 5 Classifier performance after 10 times SVM training of all 34 features |
| 表6 不同特征集分类器的表现 Table 6 Classifer performance of different characteristic sets |
目前,siRNA的设计主要有两种方法,分别是基于序列特征和基于热力学能量特征。基于序列特征的siRNA设计方法是以有效的siRNA序列特征为依据,利用统计学方法计算获得设计规则。传统的siRNA设计软件存在如下问题:①选取的样本集不同,有些方法的样本容量偏小,得到的设计规则通用性较差;②选择的特征集存在差异,得出的设计规则不尽相同,有些甚至是相悖的,缺乏统一的标准;③缺乏对特定领域的特征分析,如没有针对特定基因的GC含量特征、内部重复序列特征分析,各特征之间是独立分析的,预测精度不高,一般在50%~70%;④没有考虑脱靶效应。
从本文实验结果和对分类器的内部评估可以看出,本文方法效果较理想。文献[ 2]是本课题组2010年的研究成果,准确度为81.1%,加入本文所提的两类特征之后,分类器的预测准确度提高到88.12%。为了进一步证实本文所提出的方法真实有效,还选取了Zhi John Lu和David H.Mathews2007年的一项研究成果[ 6]作为对比,见表7。
| 表7 本文方法与文献[6]方法的结果对比 Table 7 Result comparision between reference[6] and proposed method |
从表7可以看出,两种方法的准确度相差不大,但是在敏感度方面,本文方法有较大的优越性,从敏感度的值88.35%可以看出,本文方法更接近于真实情况,真阳性率比较高。但在特异性方面,文献[ 6]中方法高了8个百分点,两种方法各有优缺点。
提出了一种基于siRNA-mRNA结合热力学特征的高效siRNA筛选方法,与传统的一些siRNA干扰效率预测方法相比,在准确度和敏感度的指标上有所提高。本文提出的将RNAup预测的siRNA-mRNA结合热力学特征应用到siRNA干扰效率预测中,希望也为以后siRNA领域的工作者提供一个全新的思路。另外,本文所提出的一种全新的3-mer编码方式被证实与siRNA干扰效率是相关的,能为以后的siRNA预测工作提供参考。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|