作者简介:王刚(1981-),男,讲师,博士.研究方向:机器学习.E-mail:wanggang.jlu@gmail.com
提出一种二阶段并行基因选择方法(TPM),可以获得最优特征子集。针对以往算法易于陷入局部极值的不足,提出了一种模糊多种群粒子群(FMP),可以有效地扩展搜索空间。通过在leukemia、colon、breast cancer、lung carcinoma、brain cancer五个数据集上的测试,验证了本文方法不仅可以获得更优特征子集和更高的分类精度,而且可以选择尺寸更小的特征子集。本文的研究成果可为基因表达领域提供一种新的思路。
A two stage parallel gene selection method (TPM) for obtaining the optimal feature subset is proposed. A fuzzy multi-swarm particle optimization (FMP) is also proposed to extend the searching spaces, to overcome the problem of traditional algorithm to be locked to local optimum. The performance of the TMP is evaluated on five microarray datasets (leukemia dataset, colon dataset, breast cancer dataset, lung carcinoma dataset and brain cancer dataset). The comparison results show that the proposed method not only gets better quality of feature subset and higher classification accuracy, but also generates smaller feature subsets. The results of this study could provide a new idea to the field of gene expression.
微阵列(Microarray)是后基因组时代最具代表性的技术之一,可以同时检测成千上万个基因在生物体内的活性,可以揭示基因表达的变化、基因间互相关系等,为疾病诊断、致病机理的研究、DNA测序、基因多位点及突变检测、药物的研制提供重要的基础。微阵列数据具有小样本、高维度、高噪声、高冗余、样本分布不均匀的特点,为研究带来了很大的困难。高维数据降维技术是有效处理微阵列数据的方法之一。
降维技术通常分为特征选择和特征抽取两类。特征选择是从原始的特征中选择一些特征形成特征子集,其中的一些特征被抛弃,一些特征被选择,被选择的特征能保留原来的物理含义。特征选择的一般过程包含产生子集、子集评估、终止标准、结果验证4个部分。特征选择方法的分类标准很多,目前普遍采用的方法是根据学习算法在计算流程中的位置作为分类标准,可分为包裹、过滤、混合3种。包裹方法中学习算法完成对特征子集的评价;过滤方法中学习算法与特征评估是完全独立的;混合方法为包裹和过滤的混合方法。为了更清晰地描述本项目的研究目的,本文根据子集的产生方式将特征选择的研究现状分为两类:基于搜索的特征选择方法和基于评价标准的特征选择方法。根据搜索的方式,目前的研究主要分为基于贪婪、启发的搜索方法和基于优化算法的搜索方法两类。前者主要依靠启发信息引导搜索的方向,后者以解决最优化问题为目的,具有随机性、全局性的特点。
通常基于启发的搜索方法可以获得更优的特征子集。遗传算法(GA)[ 1]、粒子群(PSO)[ 2]、蚁群(ACO)[ 3]是最具代表性的启发搜索方法。Yildiz[ 4]等提出了一种基于数据融合和GA的基因选择方法,在breast cancer数据集上获得了较好的实验结果。Mohamad[ 5]等通过给出粒子更新的规则,提出了一种新的离散PSO,可针对微阵列数据获得更小的特征子集。Chuang[ 6]等提出了一种PSO/GA混合算法,可获得较小的基因子集和错误率。Garcia-Nieto[ 7]采用并行粒子群在4个癌症数据集上完成了测试,获得了较优的特征子集。Abdi[ 8]等提出一种两阶段的微阵列数据分类方法,第一阶段采用mRMR去除冗余的基因,第二阶段采用PSO完成特征选择,同时用PSO优化SVM的核函数,获得了良好的分类精度。Kabir[ 9]等改进了ACO的信息素更新和启发信息度量,从而提高了算法的泛化能力,在微阵列数据集上体现出良好的性能。Li[ 10]等提出了ACO-S,包含两个阶段,提高了ACO的搜索能力,在多个微阵列数据集上得到了较好的测试结果。
综上所述,基于启发搜索的方法可在微阵列数据集上获得较好的分类精度和较短的特征子集长度,但由于微阵列数据的小样本、高维度、高噪声、高冗余、样本分布不均匀的特点,基于启发搜索的方法常常会迷失搜索方向,放弃更好的指引路径,陷入局部极值,从而获得次优的解集。本文针对搜索方向难以指引的难题,提出一种模糊多种群粒子群(FMP),显著扩大搜索范围,从全局找到更优的路径。针对高维数据,提出了一种2阶段并行基因选择方法(TPM),结合信息增益(Information gain,IG)[ 10]、FMP、SVM[ 11]完成特征选择。
FMP由多个模块构成,包括种群调控模块、种群模块、个体。其中种群调控模块完成对多个种群收敛的调节,同时也是种群间通讯的媒介。种群模块代表一个完整的种群,协调其内部个体的速度、位置等参数的更新。个体包含粒子的位置、速度等信息以及迭代质量信息。
每个种群独立地完成其内部粒子的更新,种群的结构与更新方式与binary PSO[ 12]相同,更新过程描述如下:
式中:
式中:
式中:函数
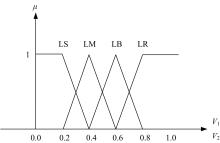
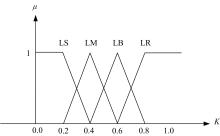
模糊逻辑控制输入和输出的论域定义为:小(LS),中(LM),大(LB),较大(LR)。图1为输入参数的隶属度函数,
式中:
| 表1 个体数量比的模糊规则 Table 1 Fuzzy rules for individual ratio |
FMP以并行计算方法描述如下:
Step1 初始化种群模块数量、种群模块中个体数量、个体位置、个体速度等参数。
Step2 并行for(当前迭代值<最大迭代值and全局最优适应度值25个迭代未更新)。
Step3 并行for(全部种群模块)。
Step4 更新种群模块位置。
Step5 更新种群模块速度。
Step6 个体计算适应度。
Step7 更新全局最优适应度和局部最优适应度。
Step8 结束并行for。
Step9 种群调控模块调节种群模块数量。
Step10 更新每个种群模块、个体的迭代质量。
Step11 if种群模块数量增加,then创建新的种群模块并销毁最差种群模块5%的个体。else销毁相应数量最差的种群模块并增加最优种群模块5%的个体。
Step12 结束并行for。
TPM包含两个阶段:第一阶段采用IG、FMP、SVM过滤冗余的基因;第二阶段采用FMP与SVM完成特征选择。
采用SVM分类正确率和IG评估设计FMP的目标函数去除冗余的基因。目标函数为:
式中: θa为SVM分类正确率的权重;
采用SVM的分类精度、迭代信息构造TPM的目标函数。目标函数为:
式中:
Step1 初始化FMP。
Step2 以并行方式执行FMP,采用式(7)作为目标函数。
Step3 获得当前微阵列数据的特征子集 M。
Step4 将 M作为第二阶段的核心特征子集,即将 M作为目标特征子集的子集。
Step5 初始化FMP。
Step6 以并行方式执行FMP,采用式(8)作为目标函数。
Step7 获得最优特征子集。
本文采用最典型的leukemia data set、colon data set、breast cancer data set、lung carcinoma dataset、brain cancer data set五个高维数据集[ 10]进行测试,覆盖的样本范围为38~203、基因数量为2000~12 600、分类数量为2~5,见表2。
| 表2 实验采用的微阵列数据集 Table 2 Microarray data sets used in the experiments |
参与比较的算法为GA、ACO、PSO、SA、TPM。其中GA参数的设置为:迭代次数=200、种群规模=40、交叉率=0.7、变异率=0.02。ACO的参数设置为:迭代次数=200、种群规模=40、 β=5、 ρ=0.1。PSO的设置为:迭代次数=200、粒子数=40、 ω=0 .9、 c1 =2、 c2=2。GA和PSO的目标函数均设置为分类精度。TPM的参数设置为:迭代次数=200、种群数量=4、每个子种群的粒子数=40、 ω=0 .9、 c1 =2、 c2=2。在目标函数(7)中, θa=0.8, θb=0.1, θc=0.1。在目标函数(8)中, θa=0.9, θb=0.1。表3为5种搜索算法在5个数据集上的分类精度测试结果,TPM获得的特征子集的分类精度均优于其他4种算法。平均分类精度的指标同样可以体现出TPM以82.37的精度明显优于其他算法。这是由于TPM多种群协作机制显著扩大了搜索范围,使得种群向正确的目标探索,获得更好解集的概率明显优于其他算法。GA以79.85位居第二。SA、PSO、ACO的性能比较接近。
| 表3 基于GA、ACO、PSO、SA、TPM的方法在 5个数据集上的分类精度对比 Table 3 Classification accuracy rate comparison between GA,ACO,PSO,SA and TPM on five data sets |
表4为5种搜索算法获得最优特征子集的长度。TPM在5个数据集上获得了最短的特征子集长度,平均特征子集长度103明显低于其他方法。由此可见,TPM不仅可以获得最高的分类精度,而且可以获得最短的特征子集长度。从这两个指标可以体现出TPM可适应多个数据集,并较为稳定。
| 表4 基于GA、ACO、PSO、SA、TPM的方法在5个数据集上获得的特征子集长度 Table 4 Selected feature subsets obtained by GA,ACO,PSO,SA and TPM on five data sets |
表5为TPM采用5种分类器的分类精度结果。采用SVM在leukemia、colon、breast cancer数据集上获得了最高的分类精度。采用kNN在Lung carcinoma数据集上获得了最高的分类精度。采用DT在Brain cancer数据集上获得了最高的分类精度。结果表明:分类器会受到数据分布的影响,无法获得较高的稳定性。SVM适用于大部分数据集,而lung carcinoma和brain cancer数据集需要采用不同的分类器才可以获得更高的分类精度。
| 表5 采用不同分类器的TPM方法在5个数据集上的分类精度 Table 5 Classification accuracy rate for TPM using multiple classifiers on five data sets |
表6为TPM采用不同分类器获得的特征子集长度。SVM在4个数据集上获得了最短的特征子集长度。DT在Breast cancer数据集获得了最短的特征子集长度。因此,根据特征子集的长度判断出SVM可较稳定地完成降维任务。
| 表6 采用不同分类器的TPM方法在5个数据集上获得的特征子集长度 Table 6 Selected feature subsets for TPM using multiple classifiers on five data sets |
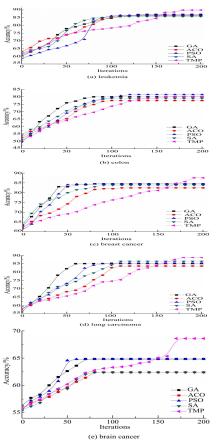
图3为5种方法分别在leukemia、colon、breast cancer、lung carcinoma、brain cancer五种数据集上的分类精度。由图可见,其他4种方法收敛速度快,但在100步附近便会陷入局部极值。TPM收敛速度较慢,可跳出局部极值,从而获得更优的解集。
充分利用PSO、SVM、模糊逻辑控制、IG、并行计算等方法提出一种二阶段并行基因选择方法(TPM),通过2个阶段的计算,完成对微阵列数据的降维和分类。针对微阵列数据的小样本、高维度、高噪声、高冗余、样本分布不均匀的特点,提出一种模糊多种群粒子群(FMP),以搜索更广阔的空间,避免过早收敛,陷入局部极值。通过在5种典型数据集上的测试验证了TPM的稳定性。通过与GA、PSO、ACO、SA四种典型方法的比较可知,TPM获得的特征子集质量高于其他4种方法,即分类精度、特征子集的长度均优于其他方法。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|