作者简介:燕杨(1981-),女,博士研究生.研究方向:模式识别与图像处理.E-mail:yan_yang10@mails.jlu.edu.cn
提出了一种基于层叠条件随机场的中文病历命名实体识别新方法,该方法在第一层条件随机场模型中实现对病历中身体基本部位或组织和基本疾病名称的识别,将识别结果传递到第二层条件随机场模型(Conditional Random Field,CRF),同时定义一个由词性和实体特征结合而成的组合特征,与字符特征、词边界特征及上下文特征共同作为第二层CRF模型的特征集,为疾病名称和临床症状两类命名实体的识别提供决策支持。在利用CRF++进行的开放测试中,本文模型相比于无自定义组合特征的层叠CRF模型,F值提高了3%;相比于单层CRF模型,F值提高了7%,总体性能有显著提高。
A new method for named entity recognition in Chinese medical records based on cascaded Conditional Random Fields (CRFs) is proposed. The first layer of the cascaded CRFs is used to identify the basic named entities of body parts and diseases. Then, the identified results are fed to the second layer for recognition of nested named entities for complex diseases and clinical symptoms. A new combination feature, composed of part-of-speech features and named entity features, is defined. This new feature together with the character features, word boundary features and context features in a sentence are taken as the feature set of the second layer. In the experiments based on CRF++, the proposed method yields a 3% higher F-score than cascaded CRF without the combination feature. Moreover, compared to single layer CRF method, it yields a 7% higher F-score, a significant increase in overall performance.
相对于通用领域,医学领域命名实体的特点使该领域中命名实体识别问题更加复杂:命名实体构词模式复杂,大量命名实体具有嵌套结构[ 1];非结构化文本缺乏统一的表述标准,命名实体之间存在歧义性;传统的特征提取方法不能利用医学概念内部的约束去获得更充分的特征信息。中文病历文本的命名实体同样具有上述特点。相关研究表明,常用的命名实体识别方法在中文病历命名实体识别领域得不到在其他通用领域所能获得的高性能[ 2]。例如,文献[3]中命名实体识别在中文新闻专线领域达到了95%,而近期文献[4]中关于中文病历命名实体识别的准确率为86.16%。
目前命名实体识别方法主要有三种:基于词典的方法、基于规则的方法和基于机器学习的方法[ 5, 6],其中,基于机器学习的方法具有更好的实用性和可移植性[ 7]。常用的机器学习模型包括支持向量机模型(Support vector machine,SVM)、隐马尔可夫模型(Hidden markov model,HMM)、最大熵马尔可夫模型(Maximum entropy markov model,MEMM)和条件随机场模型(Conditional random field,CRF)等。文献[8]的实验表明:SVM在医学文本的命名实体识别中性能不如CRF模型,因为后者能更好地利用句子的序贯状态及其与特征间的依赖关系;HMM中观测元素之间具有独立性假设,不能充分考虑上下文特征[ 9],而中文病历文本中的观测序列具有多个相互作用的特征,且观测元素之间具有依赖关系,所以HMM在一定程度上限制了特征的选择;MEMM解决了隐马尔可夫的问题,可以任意选择特征,但其只在局部做归一化,容易陷入局部最优,出现标注偏置问题[ 10];CRF模型具有表达元素之间长距离依赖性和交叠性特征的能力,能够容纳任意上下文信息,针对病历文本特点可任意设计特征集,并且它不在每一个节点进行归一化,而是对所有特征进行全局归一化,可以求得全局最优解,解决标注偏置问题[ 11],从而能够更准确地进行命名实体识别。
在医学命名实体识别领域,有相关研究证明了该模型的有效性。例如:Mc-Donald等[ 12]结合词形特征、字典特征等多种特征集合进行医学文本命名实体识别,总体F值达到82.4%;Leaman等[ 13]对医学文献中的多类实体进行识别,总体F值达81.96%;在中医学领域,Wang等[ 14]对中医病案中的病症进行智能识别,总体F值达到62.829%,这些实验都证明了CRF模型可以有效地识别医学文本中的命名实体。
如上所述,CRF模型在命名实体识别及序列标注问题中具备很好的性能。但在针对较复杂的实际应用时,如既要识别出实体的边界又要识别出实体的类型,采用单层的条件随机场模型往往很难满足要求[ 15]。因此,本文提出了一种基于层叠条件随机场(Cascaded conditional random field,CCRF)模型的中文病历命名实体识别新方法,根据中文病历中命名实体的特点,定义了一个由词性特征和实体特征结合而成的组合特征加入到模型中,实验结果表明,与单层CRF模型和无自定义组合特征的层叠CRF模型相比,CCRF模型的识别性能有明显提高。
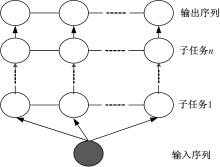
条件随机场可被定义为一个无向图模型,最早由Lafferty和McCallum在2001年提出[ 11]。如 图1所示,它是由一个点序列所表示的随机变量 Y、由边表示的条件依赖关系以及条件 X所组成,随机变量只与其相邻元素有条件依赖关系,因此,随机变量服从马尔可夫(Markov)属性,并形成一个线性(Markov)马尔可夫链。
给定输入序列 X后,标记序列 Y的条件概率可被定义为:
式中: f是整体特征向量, f中每一个元素是任意的特征选择函数 fm, m∈[1, M], M为特征函数的数量; λ是权值向量,通过模型训练获得相应的估计值; Z( X)是归一化因子,是所有可能的标签序列 S的总和:
对于输入序列 X,最有可能的输出标记序列 Y为:
层叠条件随机场模型的整体框架是一个“转移学习”的过程,即将一个复杂任务分为几个相对简单的子任务,每一个子任务对应于层叠条件随机场模型中某一层。模型从“旧的”子任务中学习,然后将结果传递给有更具体要求的新建任务,这一转移过程可实现更少的数据传输,减少信息冗余[ 15]。层叠条件随机场模型如 图2所示。
在层叠条件随机场模型中,高层模型的输入变量不仅包含观察值,还包含来自低层模型的识别结果,从而为高层条件随机场模型的识别提供决策支持。而低层模型所产生的错误可以经过适当的过滤和调整,再将结果传递到高层模型,可以避免错误的传播和扩散[ 16]。
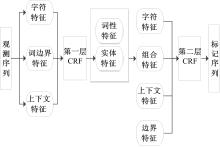
病历文本中的疾病名称和临床症状对疾病的预防与诊断起着重要作用,因此本文将对这两类命名实体进行识别研究。在翻阅了大量的病历并进行分析后,发现:①大多数的疾病名称都是由身体部位名称与基本疾病名称组合而成,如:“脑梗塞”是由“脑”与“梗塞”组成。②大多数的临床症状都是由身体部位名称与形容词或动词等具有固定词性的词组成,如“四肢无力”是由身体部位“四肢”与形容词“无力”组成。基于以上特点,本文低层模型仅以观察值为条件,进行身体基本部位或组成和基本疾病名称两类简单命名实体的识别,将识别的结果传递到高层模型,同时加入一个自定义的组合特征,它由词性和实体特征组合而成,与字符特征、词边界及上下文特征共同作为高层模型的特征集,这样高层模型的输入变量将不仅包含观察值,而且包含了来自低层模型的识别结果,从而为高层条件随机场模型对复杂疾病名称及临床症状的识别提供了决策支持,本文提出的基于层叠条件随机场模型的中文病历命名实体识别框架如 图3所示。
2.2.1 第一层CRF模型特征集选取
在本文的层叠条件随机场模型中,第一层CRF模型的观测序列以单独的一个字为token,每个病历文本都由token序列表示。每个标记直接依赖于与其对应的当前token以及窗口内相邻的其他token。这一层使用由字符特征、词边界特征以及窗口长度为5的上下文特征组合而成的特征集,具体描述如下:
(1)字符特征:本文所指的字符,包括汉字、标点符号、外文字母和数字等。
(2)词边界特征:采用BIOES编码模式来表示词边界特征(B—开始,I—内部,E—结束,O—非实体,S—单字实体),相对于BIO编码模式,BIOES编码模式能够表示更多的信息,识别效果优于BIO编码模式[ 17]。
(3)上下文特征:上下文特征是指窗口长度内token之间的相互依赖关系。在第一层CRF模型中,窗口长度设定为5,显示了窗口内相邻的5个token之间的依赖关系。
2.2.2 第二层CRF特征集选取
在第二层CRF模型中,依据中文病历中命名实体的构词特点,自定义了一种组合特征,即由词性和实体特征组成,与字符特征、词边界特征、上下文特征共同组成第二层CRF模型的特征集,具体描述如下:
(1)组合特征:该特征由词性特征与实体特征组合而成,可表示出疾病名称与临床症状的内部依赖关系,即构词特点。其中实体特征为第一层CRF模型识别后的输出结果,词性特征由中科院ICTCLAS分词器得出。
例如,在“因/p 脑/S-BC 梗/B-BD 塞/E-BD 致/a 肢/B-BC 体/E-BC 活/v 动/v 笨/a 拙/a”中,BC(身体部位)与BD(基本疾病名称)即为实体特征;p(介词)、v(动词)及a(形容词)即为词性特征,在本文模型中,实体特征与词性特征不作为独立特征存在,而是捆绑为一个整体特征,即组合特征。
(2)上下文特征:第二层CRF模型的窗口长度选择为7,显示了窗口内相邻的7个token之间的依赖关系。
本文选用中科院的ICTCLAS分词器作为分词工具,由于它不是专门面向医学领域的分词工具,缺乏专业词汇,因此我们对其进行扩展,将ICD-10' International Classification of Disease,10th Revision和ICD-9-CM2 The International Classification of Diseases,9thRevision,Clinical Modification中的术语添入其中。随机抽取来自临床医院脑血管科室的65份电子病历,构建一个共计81453个字符的语料库。分词后对病历中实体类别标识情况如 表1所示。
| 表1 本文所识别的命名实体类别 Table 1 Named entity types we recognized |
本文将一维特征模板表达为 Fn, Sn,其中字母 F, S分别对应于标注语料中的第1、2列,数字0表示当前token,非零数字表示与当前token相对的行数。 表2给出了本文实验中的一组模板文件。
| 表2 部分特征模板的设计 Table 2 Template design |
本文将多维特征模板表述为 Fn-1 /Fn, Sn-1 /Sn/Sn+1等形式,其中前者表示特征模板中第一列相邻token之间的二元特征联合,后者表示第二列相邻token之间的三元特征联合。
本文利用中科院ICTCLAS2011(http://www.ictclas.org/)分词器进行分词,使用CRF++0.53(来自http://crfpp.googlecode.com/svn/trunk/doc/index.html)开源工具对模型进行训练和测试,利用conlleval.pl(来自http://www.cnts.ua.ac.be/conll2000/chunking/output.html)程序进行评测。开放测试部分实验结果如 表3、 表4、 表5所示。
由 表3可以看出:①语料库可以标注为单字和词两种形式,这是由于对于同样多的训练语料,粒度较词小的字,可以得到更多的语料数,这对于召回率的提高有很大帮助,并且可以得到更多反映实体结构的特征,能够解决部分数据稀疏的问题,因此本文实验采用字粒度形式。②使用本文自定义组合特征的识别效果要优于普通的特征组合。这是由于本文自定义组合特征能更直接地表示出存在歧义的实体内部结构的不同,从而能够进行准确识别。自定义组合特征避免了分开使用词性特征与实体特征造成的特征信息冗余与识别混淆。如 表4所示:“头痛”,词性特征标注为“a a”,实体特征标注为“T O”,而自定义组合特征标注为“T a”;“正常”,词性标注为“a a”,实体特征为“O O”,而自定义组合特征标注为“a a”,可以看出加入自定义组合特征的方法后, F值提高了3%,而相对于单层CRF模型,则显著提高了7%。
| 表3 不同条件下的识别效果对比 Table 3 Comparison of recognition effect under different conditions |
| 表4 自定义组合特征与普通特征组合的标注 Table 4 Labels of custom combination features and common features combination |
| 表5 不同窗口长度识别效果对比 Table 5 Comparison of recognition effect with different window lengths |
由于病历文本中的疾病名称的长度基本为2-7,临床症状的长度基本为2-9,当上下文窗口长度为7时,既可以避免上下文依赖不足,又可以避免过长的窗口长度造成特征冗余,识别结果最优(见 表5),因此,本文实验中窗口长度设置为7。
在实验中,还发现,使用本文模型,即使训练文本中未出现的实体在测试中也可以被准确识别,这也是多数模型难以提供的信息。例如,训练样本中的“脑血栓”被标记为D,而“心梗塞”在训练样本中并未出现,但由于它与“脑血栓”构词模式相同,因此本文模型利用自定义组合特征,在测试文件中依然可以准确识别出“心梗塞”为D;但并不会将所有具有相同构词模式的词都标记为相同实体,例如“鼻唇沟浅”是由身体基本部位“鼻唇沟”与形容词“浅”所组成,被标记为P,而“腰间盘突出”同样也是由身体基本部位与形容词所组成的实体,但并没有被同样标记为P,而是根据上下文特征被准确地标记为D,这些方面都体现出了本文模型特有的优越性。
本文针对中文病历文本的特点,提出了一种基于层叠条件随机场模型的中文病历命名实体识别新方法,第一层CRF模型对身体基本部位或组成和基本疾病名称两类简单命名实体进行识别,将识别的结果传递到高层模型,同时加入一个本文自定义的组合特征,这样高层模型的输入变量将不仅包含观察值,还包含来自低层模型的识别结果,从而为复杂疾病名称及临床症状的识别提供了决策支持。通过对真实语料的封闭与开放测试证明,本文模型相比于无自定义组合特征的层叠CRF模型,F值提高了3%,相比于单层CRF模型, F值提高了7%,总体性能有显著提高。由本文实验可见,对于训练语料中没有出现的实体在测试中也能够被准确地识别,本文模型有效地解决了中文病历中命名实体具有嵌套结构且存在歧义性的问题。后续工作中,将对实体关系及其他重要信息进行提取,为临床决策提供更可靠的支持。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|