作者简介:董悦丽(1973-),女,教授.研究方向:计算机网络,药物分子设计.E-mail:dongyueli@neusoft.edu.cn
针对药物分子对接任务,基于传统动态任务迁移方法,提出了药物分子对接任务迁移优化策略。通过分析任务动态迁移涉及的各结点自身中断事件发生次数的数学期望和方差,得出了各结点的即时可靠性评价参数,选取可靠性高的目标结点机。在源结点和可靠的目标结点之间实施增量数据预迁移,缩短了停机拷贝时间。最后的实例测试表明,药物分子对接效率和可靠性得到了提高。
In this paper an optimization strategy of dynamic task migration for molecule docking is proposed based traditional task migration method. The mean and variance of the number of sequential jobs of each node are analyzed to obtain the coefficient of variation reliability, thus, to increase the efficiency and security of molecule docking tasks. An incremental data migration is carried out from the source node to a high reliable target node, and the time of downtime copy is shortened. The results of case study show that the efficiency and security of molecule docking tasks are improved.
随着人类基因组计划的完成、结构生物学和蛋白质纯化技术的快速发展, 适合于受体分子的靶标急剧增加。同时, 商业小分子数据库也在不断更新。这就为利用计算技术进行优化提供了空间和必要性。计算机辅助药物分子设计[1]可以对分子对接对象集进行聚焦, 也就是进行药物分子筛选, 包括预处理、分级打分等环节。常用的计算机辅助药物设计方法有:定量构效关系(Quantitative structure activity relationship, QSAR)方法、药效团(Pharmacophore)模型方法、分子对接(Molecular docking, MD)方法等。然而, 药物分子对接所对应的搜索空间非常巨大, 即便只考虑配体小分子的柔性的同时假定受体大分子为刚性的情况, 粗略估计对接所涉及的搜索空间也至少包含1030个解, 需要耗费大量的计算时间。各类计算机及优化技术大都集中在算法优化方面, 如:蒙特卡罗算法[2]、模拟退火算法[3]、遗传算法[4]等大量的智能优化算法被应用到分子对接软件中。目前, 比较成熟的分子对接软件DOCK[5]对接一个小分子的计算时间需要1~3 min左右, 如果要对包含235 000个小分子的分子库进行筛选, 即使假设1 min对接一个小分子, 也需要3917 h(约163天)。
分子对接应用面临高计算强度和快速计算的挑战。主流的分子对接软件可以运行在大型机、工作站或集群, 但由于计算资源昂贵, 因此在不同地理位置分布的、不同机型的虚拟计算资源构成的网格环境, 在药物分子对接领域得到广泛应用[6]。分子对接任务具有计算量大、执行时间长的特点, 当计算结点的可靠性下降时, 有必要对结点机实施任务迁移。
考虑到分子对接的安全性、效率以及负载均衡等问题, 进行动态任务迁移优化方案研究就显得很重要。药物分子对接在线迁移要求考虑同步内存和CPU状态以及对接数据, 而在线迁移要求该同步过程中花费较短的停机时间[7], 因此如何解决较短时间内同步大量数据, 是药物分子对接在线迁移面临的一个关键问题。本文提出了任务迁移优化策略, 通过分析各结点中断事件发生次数的数学期望和方差, 计算结点的可靠性因子, 并在结点可靠性降低至临界值后实施动态增量式迁移过程。
本文在KVM[8]虚拟机平台上实现了在线迁移优化机制的原型系统, 并搭建了实验环境。通过在具有1177个小分子的分子库中寻找受体为环氧合酶-2的配体小分子, 验证了在线任务迁移优化策略的高效性和可用性。实验结果表明, 在保证计算效率的前提下, 该策略进一步提高了分子对接计算过程的安全性。
药物分子对接即配体小分子和受体(蛋白质)大分子的结合是个复杂过程, 包括受体与配体的去溶剂化、受体(主要是活性位点处的残基)与配体的构象变化和两者之间的相互作用等。在受体的活性位点(Active site)处, 配体小分子寻找其合理的取向(Orientation)和构象(Conformation)。配体和受体的相互作用是一个综合平衡的过程[9], 并且配体小分子必须取得一定构象, 以“ 适应” 受体结合部位的“ 空腔” (Cavity)形状和构象变化。
配体小分子和蛋白质大分子对接的部位是位于大分子受体靶点的某几个氨基酸残基[10]。分子对接过程主要遵循互补匹配原则:几何形状、静电作用互补匹配, 氢键、疏水相互作用互补匹配, 即锁匙原理。
在线迁移是指在整个迁移过程中, 虚拟机暂停时间非常短, 虚拟机上运行的服务能够响应用户的请求。分子对接任务的动态迁移是将某时间点的运行状态通过计算机网络在计算结点之间进行高效复制[11], 包括CPU状态、相关寄存器状态、内存状态以及外部资源设备状态等。目前, 没有针对药物分子对接领域在线任务迁移的研究。理论上存在如下两种迁移方式:预知性中断迁移[12]和动态任务迁移[13]。
(1)预知性中断迁移
相当于停机迁移, 在对接前可以对配体小分子库设置断点, 源结点机停机后, 要将其CPU各寄存器、内存以及外部资源设备的当前状态等全部复制到目的结点机中, 在目的结点机正确加载状态参数后, 启动目的结点机进行后续的分子对接任务。
(2)动态任务迁移
进行在线任务迁移, 并最大限度地缩小源结点机的停机时间。在第一轮迁移过程中, 复制源结点机的寄存器、全部内存以及外部资源设备的状态到目的结点机; 进行若干轮的相关状态数据的增量迁移; 如果剩余增量状态数据小于设定阈值或迭代次数大于预设的阈值, 终止迭代, 停止源结点机, 将剩余的全部增量状态数据迁移到目的结点机。
陈阳等[14]结合内存推送复制以及按需复制方式, 提出了基于内存混合复制方式的在线迁移机制。但没有对结点迁移时机进行判定, 也没有对迁移目的结点进行选择。本文结合源结点的状态, 通过对目的结点可靠性因子的比较, 选择合适的迁移时机并确保目标结点运行任务的可靠性和安全性。
造成结点安全性低的因素有很多, 包括设备因素、人为破坏、环境因素等。本文重点关注发生频率最高同时也是变化性最强的一种因素, 也就是由于结点处理本身(除被分配的对接事件之外)事件的频率和强度发生变化所造成的结点计算安全性下降, 从而不能在计划的时间内完成相应的分子对接任务[15]。
建立分子对接任务结点集P{P0, P1, …, Pj, …}, 其中P0为执行当前分子对接任务的源结点, P1, P2, …是可作为分子对接任务迁移的目的结点机。
为了衡量结点本身事件的频率和强度的变化, 采用中断事件出现次数的数学期望值和均方差来进行量化比较, 并得出可靠性因子, 将其加入到任务迁移策略的考虑因素之中。
结点机Pj∈ P在执行任务Ti的过程中, 若共有S(S≥ 0, S∈ I)次中断, 则任务Ti在结点Pj的执行时间为Aij=X1+Y1+X2+Y2+…+Xs+Ys+Xs+1。其中, Xi代表子任务的计算时间, Yi代表源结点本身用于自身事务执行的中断时间。令ω =X1+X2+…+Xs+Xs+1, 则ω 代表分子对接任务自身在Pj结点所需要的计算时间。
结点计算安全性的判定基于如下两种假设:
(1)分子对接任务应被分配到负载较轻的结点上。
(2)结点本身的任务到达次数符合泊松分布。
利用如上假设及M/G/1排队系统相关理论[16], 可以得到如下命题:
命题:结点的任务符合排队系统M/G/1, 中断时间到达率为λ , 分子对接任务自身所需要的计算时间为ω , 则结点自身的中断事件的发生符合参数为λ * ω 泊松分布。
其概率表述如下:
根据上述命题, 结点Pj自身中断事件发生次数的数学期望值计算如下:
式中:ρ 表示结点Pj的利用率。结点Pj自身中断事件发生次数的方差计算如下:
式中:θ 为服务参数; μ 为服务接受率参数。在M/G/1队列系统中, 可令θ =1, 则式(3)化简为:
令可靠性因子Rk, i为结点自身中断事件发生次数的数学期望和方差的二元函数, 即任务Ti在结点Pj上的即时可靠性因子可表示为:Rk, i=φ (Ek(Ti), σ k(Ti)), 且Ek(Ti)、σ k(Ti)越大, 则Rk, i值越大。
当结点Pj的利用率参数ρ =0, 即对任意一个任务Ti, 根据式(2)(4)可得出其数学期望值Ek(Ti)=ω , 方差σ k(Ti)=0时, 该结点可靠性取得最优值, 即Rk, i=0。
综合考虑各候选结点的可靠性因子和网络拥塞因素, 提出了迁移目标结点机的选取算法, 如算法1所示。
算法1 迁移目标结点机选取算法
Function Node_Selection(P, T, R0, ε )
输入:P是结点机集合; T是分子对接任务集合; R0是源结点的可靠性因子; ε 为考虑网络拥塞等因素的差值。
输出:通过判断任务Ti在结点Pj上的即时可靠性因子Rk, i以及结点Pj自身中断事件发生次数的数学期望Ek(Ti), 得到目标结点机。
{
result=P0;
R[j]← 1;
For all j∈ P do
{
For all i∈ T do
{
计算任务Ti在结点Pj上的即时可靠性因子Rk, i;
计算结点Pj自身中断事件发生次数的数学期望Ek(Ti);
}
}
For (j=1, j< =n, i++)
{
If Rj为结点集P-{P0}中可靠性最好(可靠性因子值最小)的结点 and Rj≤ R0-ε
Result=Pj;
}
Return Result;
}
对任意结点Pj∈ P{P0, P1, …, Pj, …}, 计算其可靠因子。当同时满足如下两个条件时, 向结点Pj执行任务迁移。
条件1:Rk, i为结点集P-{P0}中可靠性最好(可靠性因子值最小)的结点。
条件2:Rk, i≤ Rk, 0-ε ; 其中, Rk, 0为源结点的可靠性因子, ε 为考虑网络拥塞等因素的差值。
在药物筛选过程中, 借助计算机辅助药物分子设计软件, 可以减少用于实验环节的候选小分子集合, 大大缩短成药时间。其中, 药物分子对接软件(如:DOCK), 通过考虑与大分子对接过程中所产生的能量值, 对可能成药的小分子进行筛选。具体做法是借助能量打分值进行综合评判, 能量值越低则成药的可能性越大。通常, 对接结果将返回所有小分子按照能量从低到高的排序集合。结合实际经验, 在进行分子对接动态迁移过程中, 考虑迁移效率, 对完成对接的小分子集合, 可根据实际情况进行设置, 比如:选取能量值较低的前20%作为目标小分子集合。根据分子对接这一特点, 在数据迁移之前, 对已完成对接的小分子、正在对接的小分子和未进行对接的小分子, 分别考虑数据迁移。动态迁移算法如算法2所示。
算法2 药物分子对接动态迁移算法.
Function DynamicMigration(P0, CurrenLigandNumber, TotalNumber)
输入:源结点机编号P0, 当前正在对接的小分子编号CurrenLigandNumber, 总共需要对接的小分子数TotalNumber;
输出:目标小分子集合Result[SequenceNo, LigandNo, EnergyScore], SequenceNo是能量值序号, LigandNo为小分子编号, EnergyScore为对接产生的能量打分值;
{
Pj=Node_Selection(P, T, R0, ε ); //确定迁移目标结点机
Num=getNumberofligand(CurrenLigandNumber); //获取循环次数, 可根据需要设置
For (i=1; i< =Num; i++ )
Result[SequenceNo, LigandNo, EnergyScore]=getLigand(DockingResultSet); //从当前对接结果中, 经过能量排序得到能量值较低的小分子集合
NumberofDocking=TotalNumber-CurrenLigandNu-
mber; //目前未对接的小分子数
Increment=100; //增量单位
IncrementalDataMigration(P0, Pj, Increment, NumberofDocking, bestLigandNo); //按照小分子编号顺序迁移, 每次传递数量为Increment
Copy the CPU and memory status for the current ligand; //暂停源虚拟机, 同步脏内存页以及当前CPU状态等
Restart Pj and run docking from CurrenLigandNumber to the last ligand; //启动目标结点Pj的对接程序
Rank the EnergyScore to get the final ligand result set; //通过比较, 得到最终目标小分子集合
Return(Result) ;
}
具体动态迁移过程分为如下6阶段:
(1)确定目标结点
根据迁移目标结点机的选取算法, 综合考虑可靠性因子和网络拥塞情况, 得到目标结点机。
(2)传递中间结果
针对已经完成对接的小分子集合, 仅对能量值排序位于前20%的目标小分子集合实施在线迁移, 传递其小分子编号及能量信息, 这就避免了复杂的信息传送。
(3)增量数据预迁移
对于未对接的小分子, 首先根据编号得知未对接小分子数量, 将未对接小分子总数传送到目的主机, 再按照编号顺序实施增量式迁移。增量单位通常为10, 即一次迁移100个小分子, 避免因大量数据同步而造成停机时间过长。
(4)停机拷贝
针对正在进行对接的小分子, 实施停机迁移, 暂停源虚拟机, 同步脏内存页以及当前CPU状态等。
(5)目标结点对接执行
按照恢复后的状态启动对接程序, 继续完成计算任务。
(6)得到对接结果
目标结点对接完成后, 通过比较能量打分值, 得到最终目标小分子集合。
通过算法1, 能够选取最优的迁移目标机, 之后再借助算法2, 完成了药物分子对接任务的动态任务迁移, 这就保证了迁移后, 计算任务能够继续在目标机执行, 并结合源结点的计算结果, 得到最终的目标小分子集合。
硬件环境:7台在不同子网的宿主机结点, 结点间带宽设置为100 MB/s, 包括一台源结点机P0, 配置为Xeon E5504 CPU, 8 GB RAM, 任务迁移目的机包括6台PC机:P1, P2, P3, P4, P5, P6, 配置均为Xeon E5504 CPU, 4 GB RAM。搭建了局域网环境下的网格平台, 共3个子网, 子网1中有P0和P1, 子网2中有P2, P3, P4, 子网3中有P5和P6, 子网之间通过千兆交换机连接。
软件环境:KVM宿主机软件环境为Ubutun Server 10.04 TLS, 内核版本2.6.32-24, KVM版本kvm-88, 分子对接软件为DOCK6.2, 参数使用默认值。
实验选择的受体:选取环氧合酶-2作为受体, 它的PDB编号是4COX, 其大分子结构如图1所示。
实验使用的配体小分子库:配体小分子库中有1177个小分子。
结点性能因素:受体为环氧合酶-2时, 对接花费时间在0.1 s至12 s范围内。如果只在P0运行对接程序, 对接执行10次的平均时间为206 375 s。如果只在P1至P6运行对接程序, 对接执行10次的平均时间为247 788 s。从以上结果可以看出, 当任务从P0向目标结点(P1至P6)中的某个结点进行迁移时, 因结点性能因素, 对接执行的总体时间会增加约20%。
如果没有特别指出, 同一组应用负载下迁移实验重复10次, 本节中实验结果为10次结果的平均值。
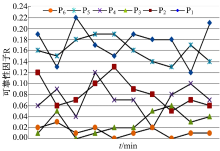
每一组迁移实验中, 当P0计算结点对接计算任务执行到100 000 s左右, 环氧合酶-2受体分子对接执行到第563号配体小分子时, 对结点P0进行大量磁盘I/O操作, 从而导致其降低性能至临界值, 触发迁移。各目标结点的可靠性因子变化趋势如图2所示。
从图2可以看出, P6结点的方差(抖动性)最小, 表示结点负载相对稳定, 同时数学期望值最小, 表示该结点负载较轻。受各目标结点负载及性能影响, 从源结点向各个目标结点的迁移时间及在目标结点对接小分子库中剩余的小分子的时间各不相同, 具体时间花销如表1所示。
| 表1 目标结点迁移时间和对接时间 Table 1 Migration and docking time of the target hosts |
综合比较可靠性因子取值及变化趋势, 选择P6结点作为目标结点实施在线迁移。在线迁移过程, 向P6结点传递目标小分子集合, 包括小分子编号及其能量值以及未对接小分子数量, 并同步内存页及CPU状态, 按照增量方式传递小分子数据, 重新启动分子对接过程。其中, 迁移所用时间为97.2 s, 迁移后, 目标计算机能够正确完成计算任务, 所用时间为120 724.6 s。这样, 利用在线迁移技术, 在P0和P6所花费对接时间, 再加上迁移所用时间为220 821.8 s。对接花费时间与目标机器单独计算花费时间接近, 总体计算效率可以接受, 并确保了分子对接计算的安全性。
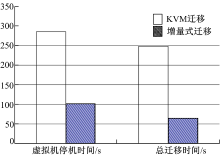
实验的最后, 我们对比了实施增量式迁移前后的迁移性能, 包括总迁移时间以及虚拟机停机时间, 实验结果如图3所示。
从图3中不难看出, 实施增量式迁移之后大幅度缩短了迁移的总体时间。采用增量式迁移在对小分子数据实施迁移时, 由于分子对接过程具有按照小分子编号顺序对接的特点, 因此以增量的方式将最近使用的小分子最早传递给目标结点, 使对接过程及早启动并持续执行, 大大提高了迁移时间。增量式迁移方式比KVM迁移平均复制数据页的迭代收敛速率高, 在停机时脏页面数较少。相反, KVM迁移机制在停机时需要复制更多的脏页面数, 因此停机时间稍长, 大约为286 ms, 大于增量式迁移中的101 ms平均时间。
通过上述对比可以看出, 在总体迁移时间和虚拟机停机时间方面, 运用在线增量式迁移机制进行药物分子对接迁移效率明显提升。
网格环境下分子对接应用计算量大, 计算时间长, 为提高其安全性及可靠性, 本文提出了分子对接过程中的动态任务迁移优化策略, 该策略考虑目标结点的稳定性和安全性, 依据方差和数学期望确定可靠性因子, 确定迁移目标结点, 从而能够使药物分子对接计算在网格环境下并行计算, 避免用户因某个计算结点负载过重或者出现网络中断、断电情况而造成的损失, 确保了药物分子对接任务的有效性和安全性。
同时, 结合药物分子对接过程的特点, 提出了药物分子对接动态迁移机制, 在选择安全稳定的目标结点后, 实施增量式数据预迁移, 从而大大提高了对接效率。
本文提出的动态任务迁移优化策略已经应用到网络环境下药物分子对接平台, 下一步, 希望能够对云计算环境动态任务迁移出现的新特性, 比如:数据安全性、多任务迁移等进行研究, 从而实现云环境下的药物分子对接任务迁移。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|